Kaggle COVID-19 CT 이미지의 분류를 딥러닝으로 수행한 프로젝트입니다. CNN 기반으로 코로나 바이러스 감염 여부를 분류하는 것이 목적이며, 이번 포스트에서는 본격적인 모델링에 앞서 OpenCV를 이용하여 이미지를 불러들이고 폴더를 나누는 작업과 기본 CNN 모델 간단 적합을 진행한다.

해당 자료는 감염자 501명과 비감염자 550명의 CT 자료로 512X512 greyscale 픽셀 이미지로 구성되어 있다. 샘플 이미지는 위와 같으며, OpenCV로 데이터를 읽어 일단 Train 70%, Validate 10%, Test 20% 비율로 나누고 폴더에 나눠 저장한다. 이는 추후에 ImageDataGenerator를 통해 데이터를 읽거나 증강할 때 편리하기 때문이다. 본래 CT 3차원이지만 이 데이터는 다양한 각도로 투영된 2차원 이미지가 섞여있다. 그리고 샘플만 보고는 감염 여부를 확인하기는 어렵다.
# 데이터 읽기 및 폴더 나누기
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import cv2
import os
def load_images_from_folder(folder):
images = []
for filename in os.listdir(folder):
img = cv2.imread(os.path.join(folder,filename), cv2.IMREAD_GRAYSCALE) # default는 512
# img = cv2.resize(img, dsize=(256, 256), interpolation=cv2.INTER_AREA)
if img is not None:
images.append(img)
return images
folder = "./COVID_CT/Not_Infected"
n_infected = load_images_from_folder(folder)
folder = "./COVID_CT/Infected"
infected = load_images_from_folder(folder)
# test(110, 100)-20%, validate(55, 50)-10%, train(395, 351)-70% 비율로 랜덤하게 나누기
ntr = round(550*0.5)
nval = round(550*0.1)
nte = round(550*0.4)
itr = round(501*0.5)
ival = round(550*0.1)
ite = round(501*0.4)
# 데이터를 저장할 상위 폴더 'COVID_CT_NEW2' 만들기
covid_dir = '/content/gdrive/My Drive/COVID_CT_NEW2'
os.mkdir(covid_dir)
train_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/train'
vali_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/validate'
test_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/test'
os.mkdir(train_dir)
os.mkdir(vali_dir)
os.mkdir(test_dir)
# 감염자 데이터를 저장할 폴더 만들기
train_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/train/Infected'
vali_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/validate/Infected'
test_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/test/Infected'
os.mkdir(train_dir)
os.mkdir(vali_dir)
os.mkdir(test_dir)
# 비감염자 데이터를 저장할 폴더 만들기
train_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/train/n_Infected'
vali_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/validate/n_Infected'
test_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/test/n_Infected'
os.mkdir(train_dir)
os.mkdir(vali_dir)
os.mkdir(test_dir)
# 감염자 이미지 저장 함수
import os, shutil
def ninfected_images_to_folder(n_infected):
train_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/train/n_Infected/'
vali_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/validate/n_Infected/'
test_dir = '/content/gdrive/My Drive/COVID_CT_NEW2/test/n_Infected/'
for i in range(len(n_infected)):
if i in range(0,ntr+1):
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(train_dir + fname)), n_infected[i])
cv2.waitKey(0)
elif i in range(ntr+1,ntr+nval+1):
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(vali_dir + fname)), n_infected[i])
cv2.waitKey(0)
else:
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(test_dir + fname)), n_infected[i])
cv2.waitKey(0)
# 비감염자 이미지 저장 함수
import os, shutil
def infected_images_to_folder(infected):
train_dir = '/content/gdrive/My Drive/자료분석특론/프로젝트/COVID_CT_NEW2/train/Infected/'
vali_dir = '/content/gdrive/My Drive/자료분석특론/프로젝트/COVID_CT_NEW2/validate/Infected/'
test_dir = '/content/gdrive/My Drive/자료분석특론/프로젝트/COVID_CT_NEW2/test/Infected/'
for i in range(len(infected)):
if i in range(0,itr+1):
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(train_dir + fname)), infected[i])
cv2.waitKey(0)
elif i in range(itr+1,itr+ival+1):
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(vali_dir + fname)), infected[i])
cv2.waitKey(0)
else:
fname = '{}.jpg'.format(i)
cv2.imwrite(os.path.join(str(test_dir + fname)), infected[i])
cv2.waitKey(0)
# 이미지 저장
ninfected_images_to_folder(n_infected)
infected_images_to_folder(infected)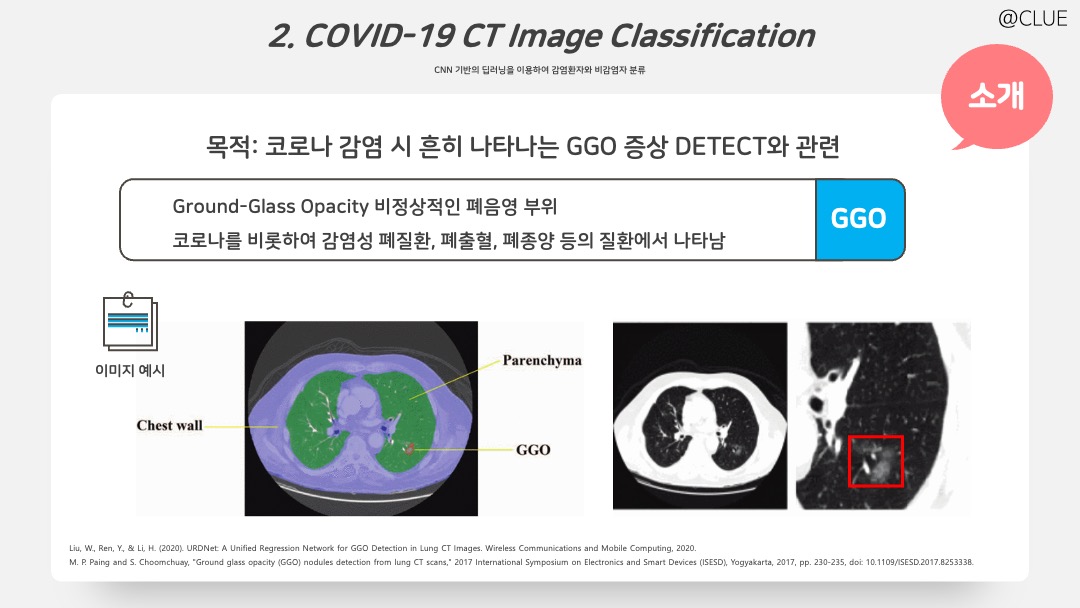
코로나 감염 시 흔하게 나타나는 증상은 GGO(Ground-Glass Opacity) 와 관련이 있으며, GGO란 비정상적인 폐음영 부위로 코로나를 비롯하여 감염성 폐질환, 기생충질환, 폐출혈, 간질성 폐질환, 폐종양 등의 질환이 CT 영상에 GGO 형태로 관찰된다. 결절성 간유리 음영으로 불리우는 GGO의 이미지 예시는 위와 같다.
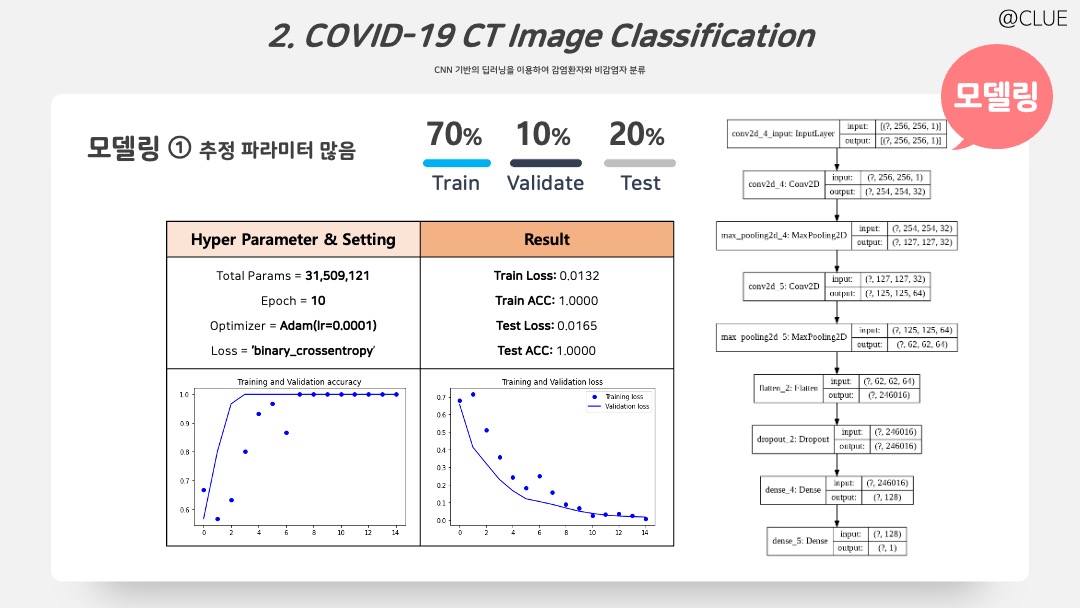
마지막으로 ImageDataGenerator를 사용하여 데이터를 256X256 픽셀로 줄여서 사용한다. 그리고 아무렇게나 쌓은 Convolution과 MaxPooling 층 두 쌍과 Dropout을 추가한 모델을 적합했다. 이는 추정해야할 파라미터가 매우 많은 모델로, Validation Accuracy 시각화를 보면 과적합이라고 보긴 어려우나, 7번 이상 반복 시 Accuracy가 1에 수렴한다.
### CNN 모델 적합 ###
IMAGE_ROWS = 256
IMAGE_COLS = 256
BATCH_SIZE = 30
IMAGE_SHAPE = (IMAGE_ROWS,IMAGE_COLS,1)
import tensorflow as tf
from tensorflow.keras import layers
from tensorflow.keras import models
model1 = models.Sequential()
model1.add(layers.Conv2D(32, (3,3), activation='relu', input_shape=IMAGE_SHAPE))
model1.add(layers.MaxPooling2D((2,2)))
model1.add(layers.Conv2D(64, (3,3), activation='relu'))
model1.add(layers.MaxPooling2D((2,2)))
model1.add(layers.Flatten())
model1.add(layers.Dropout(0.5))
model1.add(layers.Dense(128, activation='relu'))
model1.add(layers.Dense(1, activation='sigmoid'))
model1.summary()
from tensorflow.keras import optimizers
model1.compile(optimizer=optimizers.Adam(learning_rate=1e-4),
loss='binary_crossentropy',
metrics=['acc'])
history = model1.fit_generator(train_generator, steps_per_epoch=1, epochs=10,
validation_data = validation_generator, validation_steps = 1)
tr_score = model1.evaluate_generator(train_generator)
print('Train Loss : {:.4f}'.format(tr_score[0]))
print('Train Accuracy : {:.4f}'.format(tr_score[1]))
te_score = model1.evaluate(test_generator)
print('Test Loss : {:.4f}'.format(te_score[0]))
print('Test Accuracy : {:.4f}'.format(te_score[1]))### 모델 적합 시각화 ###
import matplotlib.pyplot as plt
acc = history.history['acc']
val_acc = history.history['val_acc']
loss = history.history['loss']
val_loss = history.history['val_loss']
epochs = range(len(acc))
plt.plot(epochs, acc, 'bo', label = 'Training acc')
plt.plot(epochs, val_acc, 'b', label='Validation acc')
plt.title('Training and Validation accuracy')
plt.figure()
plt.plot(epochs, loss, 'bo', label = 'Training loss')
plt.plot(epochs, val_loss, 'b', label='Validation loss')
plt.title('Training and Validation loss')
plt.legend()
plt.show()
from tensorflow.keras.utils import plot_model
plot_model(model1, to_file='CNN1.png', show_shapes=True)데이터와 모델링을 조금 더 발전시킨 내용에 대해서는 다음 포스팅을 참고바란다.
피피티 디자인 저작권은 @PPTBIZCAM
